Archive
Ekstraksi fitur Radiomics (2)
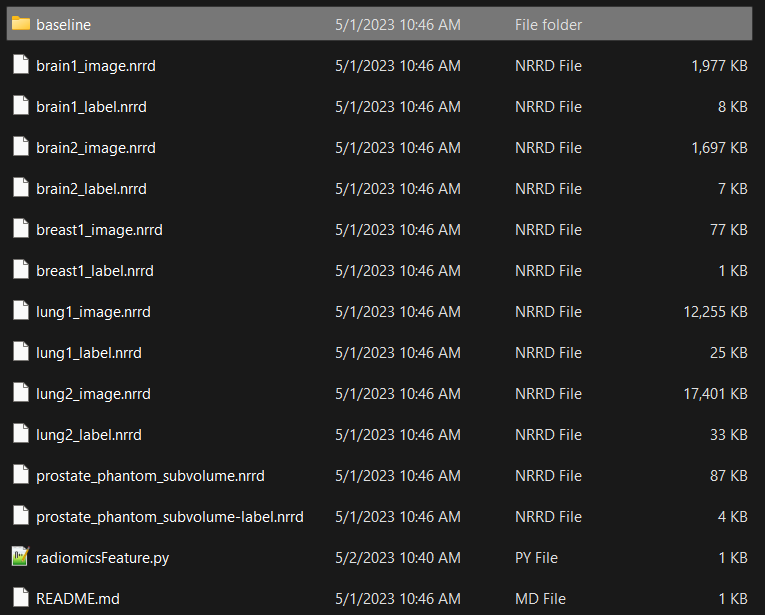
Berdasarkan penjelasan di sini, saya lalu meng-cloning data dan kode yang ada di sini. Setelah selesai proses cloning, directory yang data disebutkan berisi file seperti pada Gambar 1 berikut.
Sebuah file berkestensi py pada Gambar 1 adalah file yang scriptnya disajikan berikut ini.
import SimpleITK as sitk
import numpy as np
import os, six
from radiomics.featureextractor import RadiomicsFeatureExtractor
from radiomics import getTestCase
nama=['brain1', 'brain2', 'breast1', 'lung1', 'lung2']
for i in nama:
imageName, maskName = getTestCase('brain1', '.')
print(imageName, maskName)
extractor = RadiomicsFeatureExtractor()
result = extractor.execute(imageName, maskName)
for key, val in six.iteritems(result):
print("\t%s: %s" %(key, val))
Untuk script tersebut, file dengan konten prostate_phantom tidak terlibat karena sulit mencari hubungan antara variabel imageName dan maskName. Hubungan tersebut sangat diperlukan agar perintah execute dapat dijalankan secara otomatis. Nah, kendalanya adalah bagaimana mendapatkan kedua file tersebut (imageName dan maskName) dari DICOM.
Ekstrak fitur Radiomics
Setelah beberapa waktu saya melakukan eksplorasi DICOM, sekarang saya akan coba mempelajari cara mengekstrak fitur radiomics, gabungan fitur bentuk dan tekstur. Pustaka yang akan digunakan adalah pyradiomics. Tetapi, sebelum dapat digunakan file DICOM yang sebelumnya dipelajari harus diubah formatnya, apakah ke NifTI atau nrrd.
Dijelaskan bahwa NifTI is very explicit, with a fixed 348-byte binary header. This makes it very simple to read. However, it is a bit harder to write, because it demands the order of image data dimensions (e.g. a Red, Green, Blue image MUST be saved RGBRGBRGB…), the first three dimensions must be spatial the fourth temporal and dimensions 5,6,7 are up to the user.
Sedangkan NRRD adalah text based, so it is human readable. It is easy to write, as you can easily describe whatever order you want to your dimensions. The trade off is that it is harder to create a reader, as you need to juggle the image dimensions. What is elegant about NRRD is that the flexibility of the header allows you to create a tiny header that describes a complicated image in a different format, for example you could write a NRRD-format nhdr file that describes most uncompressed DICOM images allowing easy support for DICOM.
Untuk melakukannya, saya menggunakan panduan yang disediakan di sini dan 3DSlicer 5.2.2 pada dataset ini. Jika aplikasi 3DSlicer meminta plugin untuk diinstalasi, kita dapat melakukan instalasi melalui menu View -> Extension Manager. Untuk kasus dataset yang saya gunakan, 3DSlicer meminta dipasangkan plugin PETDICOMExtension dan SlicerRT. Konversi akan dilakukan ke format NRRD. Hasilnya seperti Gambar 1 berikut. Jika dilihat dari prosesnya, hasil konversi tersebut berasal dari 1 pasien.
Setelah itu, kita perlu membuat file konfigurasi yang tujuannya untuk mengkonfigurasi, salah satunya, fitur radiomics apa yang akan diekstrak. Detil konfigurasinya dapat dipelajari di sini. Ada empat jenis konfigurasi yang dapat disetup, masing-masing Image Types, Enabled Features, Settings, Voxel-based specific settings. Sedangkan jenis sub konfigurasi Settings juga ada empat, masing-masing Feature Extractor Level, Filter Level, Feature Class Level, Feature Class Specific Settings. Baris perintah berikut adalah contoh file konfigurasi yang perlu didefinisikan untuk mengambil fitur radiomics. Meski menurut contoh tersebut tidak disarankan membuat konfigurasi sendiri.
imageType:
Original: {}
LoG: {'sigma' : [1.0, 3.0]}
Wavelet:
binWidth: 10
featureClass:
glcm:
glrlm: []
firstorder: ['Mean', 'StandardDeviation']
shape:
- SurfaceArea
setting:
binWidth: 25
resampledPixelSpacing:Kemudian, bagaimana konfigurasi yang diperlukan untuk menyimpan fitur radiomics ke file dapat dipelajari di sini. Sayangnya, setelah mencoba beberaoa kali, saya masih gagal mengekstrak fitur radiomics karena script yang dibuat menghasilkan pesan kesalahan berikut.
raise ValueError('No labels found in this mask (i.e. nothing is segmented)!')
ValueError: No labels found in this mask (i.e. nothing is segmented)!Pesan tersebut disebabkan karena argumen kedua dari perintah execute tidak diberikan dengan tepat. Saya belum tahu label yang diminta dari mask ada di mana, harus diberikan dalam bentuk apa, dll. Tapi sepertinya, selain file berekstensi NRRD, harus ada file yang berisi label organ atau hasil segmentasi. Jadi PR berikutnya….
Eksplorasi DICOM (3)
Sebelumnya kita sudah bisa mengakses volume citra dengan perintah volread. Analisis volume citra memungkinkan kita mendapatkan informasi yang lebih banyak karena perspektif yang beragam. Ilustrasinya seperti ditunjukkan Gambar 1 yang diperoleh di sini.
Untuk mendapatkan informasi yang berasal dari gabungan bidang irisan tersebut, kita perlu memahami beberapa istilah berikut.
- Shape: menunjukkan jumlah baris dan kolom piksel pada irisan tertentu. Pada contoh yang telah digunakan sebelunnya, nilai shape adalah 44, 256, 256
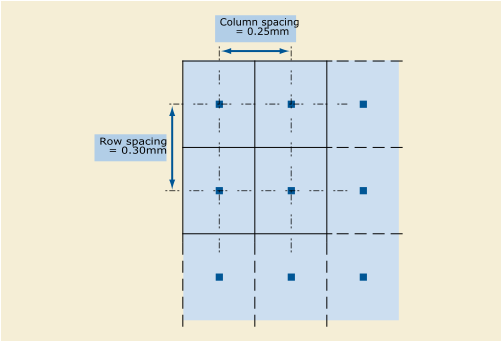
- Sampling: merupakan kombinasi dua atribut, masing-masing SliceThickness dan PixelSpacing. PixelSpacing merepresentasikan jarak fisis pada pasien dan terdiri dari pasangan nilai numerik, masing-masing adalah row spacing dan column spacing. Row spacing merupakan jarak titik tengah dari 2 baris berurutan. Demikian juga dengan column spacing yang merupakan jarak titik tengah 2 kolum yang bersebelahan. Ilustrasinya ditunjukkan di Gambar 2, yang diperoleh di sini. Sederhananya, parameter PixelSpacing merupakan sampling perangkat scanning terhadap organ tubuh pasien. Semakin kecil pasangan nilai yang direpresentasikan PixelSpacing semakin detil informasi yang diperoleh. Jarak tersebut memiliki satuan mm. Untuk contoh dataset yang sebelumnya digunakan, nilai yang diperoleh adalah sebagai berikut. Dua baris pertama menunjukkan perintah untuk mendapatkan nilainya, yang ditunjukkan pada baris ke-3. WordPress makin bagus (seharusnya), faktanya saya makin kesulitan untuk memformat tulisan di dalamnya. Soal format, LaTex memang gak ada lawannya.
d0, d1, d2 = vol.meta['sampling']print("Sampling:\n\t", "Axial=", d0, "mm\n\t", "Coronal=", d1, "mm\n\t", "Sagittal=", d2, "mm")- Sampling:
- Axial= 0.0 mm
- Coronal= 1.1719 mm
- Sagittal= 1.1719 mm
- Pixel Aspect Ratio: merupakan perbandingan antara nilai-nilai yang diperoleh sebelumnya, yaitu
d0,d1dand2. 3 baris pertama adalah formula untuk mendapatkan aspect ratio, sedangkan baris ke-4 adalah hasil perhitungannya untuk kasus yang sebelumnya digunakan. Ilustrasi dari aspect ratio disajikan pada Gambar 3 yang diperoleh di sini.axial_asp = d1/d2sagittal_asp = d0/d1coronal_asp = d0/d2- Pixel Aspect Ratio:
- Axial= 1.0
- Coronal= 0.0
- Sagittal= 0.0
- Fields of View: diperoleh dengan mengalikan parameter bersesuaian yang diperoleh dari sampling dan pixel aspect ratio. Baris pertama adalah perintah yang digunakan untuk menampilkan nilai Fields of View. Sedangkan baris ke-2merupakan hasil eksekusi dari perintah di baris pertama.
print("Field of View:\n\t", "Axial=", n0*d0, "mm\n\t", "Coronal=", n1*d1, "mm\n\t", "Sagittal=", n2*d2, "mm")- Pixel Aspect Ratio:
- Axial= 1.0
- Coronal= 0.0
- Sagittal= 0.0
Sementara saya cukupkan dulu hasil belajar kali ini. Perlu perenungan lebih panjang untuk bagian ini, terkait peruntukannya dalam analisis citra RT. Hal ini disebabkan karena:
- Slice Thickness dijelaskan sebagai simply the nominal slice thickness in mm. Belum dapat dipahami apa maksud “nominal” dalam definisi tersebut.
- Di Gambar 1 ada tiga jenis slice, masing-masing sagittal, coronal dan transverse. Sedangkan ketika membahas tentang sampling, yang muncul justru Axial, bukan Transverse. Menyamakan keduanya juga belum bisa dipastikan karena belum ditemukan pernyataan pendukung dari sumber ini.
- Contoh nilai sampling dan saya gunakan juga berbeda, khususnya slice Axial. Apakah jika sampling bernilai 0 maka detil organ tubuh diperoleh lengkap tanpa sampling, juga belum bisa dipastikan.
Selamat beristirahat….
Eksplorasi DICOM (2)
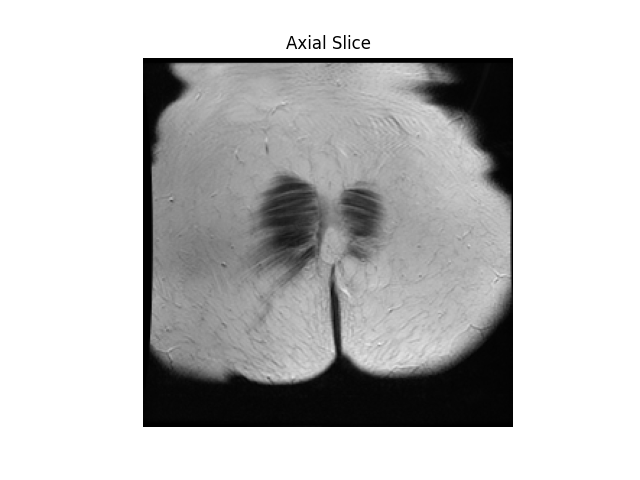
Kita lanjutkan lagi belajarnya, masih dari laman ini. Setelah kita bisa mengakses metadata yang terdapat pada sebuah file DICOM, seperti yang pada pembelajaran sebelumnya diwakili oleh variabel img, maka melalui varibel tersebutlah, sebuah citra direpresentasikan. Perhatikan Gambar 1 berikut.
Gambar 1 tersebut diperoleh dari script berikut kelanjutan dari script sebelumnya di laman ini.
from matplotlib import pyplot as plt
plt.imshow(img, cmap='gray')
plt.axis('off')
plt.title('Axial Slice')
plt.show()
Sejumlah file DICOM juga dapat dikumpulkan dalam satu directory, yang masing-masingnya mewakili satu slice atau frame dari seorang pasien. Dengan script berikut, kita bisa mengakses sejumlah file DICOM tersebut sekaligus. Sekali lagi, script ini adalah kelanjutan dari script sebelumnya di lama ini.
vol=imageio.v2.volread(lokasi[0], 'DICOM')
plt.imshow(vol[10,:,:], cmap='gray')
plt.axis('off')
plt.title('Axial Slice')
plt.show()
Variabel vol[10,:,:] menunjukkan file DICOM ke-11 dari directory tersebut. Dan Gambar 2 menunjukkan file DICOM ke-11.
Perlu diketahui bahwa nilai yang terkandung dalam matriks yang akhirnya muncul sebagai Gambar 1 dan Gambar 2 tersebut merupakan nilai Housfield Unit (HU). HU adalah nilai tak berdimensi yang umum digunakan dalam Computed Tomography (CT). Nilai HU mengacu pada koefisien atenuasi linear CT pada air dengan suhu 0 derajat Celcius dan tekanan 1 juta Pascal.
Sampai di sini dulu ekplorasinya, karena sekali lagi, waktu berbuka sudah dekat. Terlebih, beberapa hali lalu saya dibuat bingung dengan perintah volread yang harusnya menerima 2 argumen, dan argumen kedua langsung disebutkan nilainya, tidak perlu nama variabel argumennya.
Eksplorasi DICOM
Tulisan ini adalah media belajar saya terkait eksplorasi berkas DICOM (Digital Imaging and Communication in Medicine). Jika disimpan di laptop, maka pasti akan sulit mencarinya, sebab ini bukan kali pertama. Dan karena ada keperluan mendesak untuk menguasainya, jadilah sub kategori baru di buku saku ini.
Eksplorasi ini saya mulai dari sini, sebuah blog yang sejauh ini menyajikan detil informasi terkait apa itu DICOM, apa saja yang disimpan di dalamnya, dan bagaimana mengambil informasi tersebut. Selain Imaging, DICOM mengandung informasi lain yang dapat menjadi media komunikasi antar modalitis dalam infrastruktur kesehatan. Informasi tersebut dikenal sebagai metadata yang antara lain berisi:
- Atribut terkait pasien, seperti nama, usia, gender, dll.
- Atribut terkait modaliti (CT, MRI, PET, dll), seperti manufaktur, waktu akuisisi data, dll
- Atribut terkait citra, seperti bentuk, sampling, aspect ratio, pixel data, dll
Atribut lainnya dari DICOM dijelaskan di sini. Dan luar biasanya, ada banyak sekali atribut DICOM di sana.
Nah, di blog acuan, penulis menyediakan contoh yang ia simpan di sini. Sementara, saya mencoba untuk membaca file DICOM yang tersedia di sini dan sudah selesai saya unduh. Tetapi, struktur filenya berbeda. Jadi saya explore metadata.csv yang disertakan ketika pengunduhan dilakukan. Pada file tersebut, minimal ada 2 atribut penting yang bisa langsung saya pahami, masing-masing adalah File Location dan Number of Images. Dengan script berikut, saya bisa membuat titik awal ekplorasi yang sama dengan contoh di sini. Sayangnya, saya kehilangan formating untuk script yang memungkinkan kita merujuk pada baris-baris perintah karena ada penanda urutannya.
import pandas as pd
import imageio
import os
data=pd.read_csv('metadata.csv')
lokasi=data['File Location']
jumlah=data['Number of Images']
images=[]
for i in os.listdir(lokasi[0]):
images.append(i)
img=imageio.v2.imread(lokasi[0]+'/'+images[0])
img.meta
Penjelasannya adalah sebagai berikut:
- 3 baris pertama digunakan untuk meng-import pustaka
- Baris ke-4 digunakan untuk membaca file metadata.csv yang berisi keterangan tentang lokasi directroy dan file DICOM yang ada di dalamnya. Karena itu, pastikan untuk menjalankan script ini di lokasi yang sama dengan file metadata.csv berada.
- Baris ke-5 digunakan untuk mengambil kolom yang header-nya adalah File Location dan disimpan dalam variabel lokasi. Di dalam variabel lokasi yang ber-type list, terdapat daftar lokasi directory yang bisa dirujuk dengan nomor indeks.
- Baris ke-6, sama seperti perintah baris ke-5 tetapi digunakan untuk menyimpan informasi jumlah file yang terkandung di dalam directory yang bersesuaian pada kolom File Location. Ini hanya diperlukan untuk memastikan apakah benar jumlah file yang dijelaskan pada file metadata.csv sama seperti jumlah file yang tersimpan. Pada kasus ini sama.
- Baris ke-7 digunakan untuk menyimpan nama-nama file pada directory yang akan dituju sehingga dapat dengan mudah dirujuk.
- Baris ke-8 dan 9 digunakan untuk menyimpan nama file ke dalam list yang variabelnya dideklarasikan di baris ke-7
- Baris ke-10 digunakan untuk mengakses file pertama dalam list
- Baris ke-11 digunakan untuk menampilkan informasi metadata dari file DICOM pertama yang kita baca. Metadata ini tersusun dalam bentuk dictionary.
Sementara kita cukupkan sampai di sini, karena waktu berbuka sudah dekat….
Perkalian matriks dengan representasi vektor
Yang dimaksud oleh judul ini adalah operasi perkalian matriks namun semua matriks yang terlibat direpresentasi sebagai vektor. Selanjutnya, beberapa aturan yang berlaku adalah sebagai berikut:
- Matriks yang terlibat berisi nilai integer pada rentang 0 s/d 9.
- Pengguna akan diminta untuk memasukkan dimensi matriks yang akan dikalikan, masing-masing jumlah baris dan kolom matriks pertama dan jumlah kolom matriks kedua. Jumlah baris matriks kedua disamakan dengan jumlah kolom matriks pertama.
Dan berikut ini adalah scriptnya.
#include<iostream>
#include<cstdlib>
#include<string>
#include<ctime>
using namespace std;
int perkalian1(int* x, int* y, int* z, int rows, int cols1, int cols2){
int temp=0;
int l=0;
for(int i=0;i<cols2;i++) {
temp=0;
for(int j=0;j<rows;j++) {
temp=0;
for(int k=0;k<cols1;k++){
temp=temp+(x[(i*cols1)+k]*y[(k*cols2)+j]);
}
z[l]=temp;
l=l+1;
}
}
return 0;
}
int main(int argc, char* argv[]){
int rows, cols1, cols2, min, max;
max=9;
min=0;
srand(time(0));
cout << "Matrix #1" << endl;
cout << "The number of rows: ";
cin >> rows;
cout << "The number of columns: ";
cin >> cols1;
cout << "Matrix #2, its rows should equal to Matrix #1 columns" << endl;
cout << "The number columns: ";
cin >> cols2;
int sizeX=rows*cols1;
int* x=(int*)malloc(sizeX*sizeof(int));
int sizeY=cols1*cols2;
int* y=(int*)malloc(sizeY*sizeof(int));
int sizeZ=rows*cols2;
int* z=(int*)malloc(sizeZ*sizeof(int));
if(!x) {
cout << "Memory allocation for x failed!" << endl;
exit(1);
}
if(!y) {
cout << "Memory allocation for y failed!" << endl;
exit(1);
}
for (int i=0;i<sizeX;i++) {
x[i]=rand() % (max-min+1) + min;
}
int k=0;
for (int i=0;i<rows;i++) {
for (int j=0;j<cols1;j++) {
cout << x[k] << " ";
k=k+1;
}
cout << endl;
}
cout << endl;
for (int i=0;i<sizeY;i++) {
y[i]=rand() % (max-min+1) + min;
}
k=0;
for (int i=0;i<cols1;i++) {
for (int j=0;j<cols2;j++) {
cout << y[k] << " ";
k=k+1;
}
cout << endl;
}
cout << endl;
perkalian1(x,y,z,rows,cols1,cols2);
k=0;
for (int i=0;i<rows;i++) {
for (int j=0;j<cols2;j++) {
cout << "z[" << i << "][" << j << "]=" << z[k] << " ";
k=k+1;
}
cout << endl;
}
free(x);
free(y);
free(z);
}
Matriks dengan representasi vektor
Yang dimaksud oleh judul tersebut adalah menyimpan vektor (berdimensi lebih besar atau sama dengan 2) dengan representasi vektor (berdimensi 1). Matriks yang dimaksud dapat berujud seperti Gambar 1 yang diperoleh dari sini.
Script akan dibuat dalam C++ dan mengharuskan pengguna memberikan argumen berupa
- ukuran baris,
- ukuran kolom,
- ukuran lebar,
- nilai minimum dan
- nilai maksimum
Nilai minimum dan maksimum diperlukan untuk membatasi bilangan integer yang dihasilkan melalui fungsi random. Berikut adalah script yang akan mensimulasi pekerjaan tersebut. Perlu diperhatikan, saat pengolahan matriks (termasuk menampilkannya ke pengguna melalui layar monitor), kita perlu susunan bilangan yang kita maksud sudah sesuai posisinya, seperti ilustrasi di Gambar 1.
#include<iostream>
#include<cstdlib>
#include<string>
#include<ctime>
using namespace std;
int main(int argc, char* argv[]){
int rows, cols, width, min, max;
if (argc==6) {
rows=stoi(argv[1]);
cols=stoi(argv[2]);
width=stoi(argv[3]);
min=stoi(argv[4]);
max=stoi(argv[5]);
}
srand(time(0));
int size=rows*cols*width;
int* x=(int*)malloc(size*sizeof(int));
if(!x) {
cout << "Memory allocation failed!" << endl;
exit(1);
}
for (int i=0;i<size;i++) {
x[i]=rand() % (max-min+1) + min;
}
cout << "Matriks X" << endl;
int l=0;
for (int i=0;i<width;i++) {
cout << "Layer-" << i << endl;
for (int j=0;j<rows;j++) {
for (int k=0;k<cols;k++) {
cout << "x[" << j << "][" << k <<"]=" << x[l] << " ";
l=l+1;
}
}
cout << endl;
}
free(x);
}
Menyiapkan matriks dalam C++
Karena diminta untuk mengerjakan project dalam C++, maka saya harus kembali belajar dari awal. Kali ini, saya akan menyiapkan matriks yang ukuran dan batasan nilainya ditentukan oleh pengguna. Keterbatasannya adalah program berikut ini hanya mengasumsikan bahwa pengguna sudah mengetahui bahwa program yang akan digunakan menghasilkan matrik bernilai integer. Kemudian pengguna juga sudah mengetahui bahwa urutan argumen yang harus diberikan adalah jumlah baris, jumlah kolom, nilai minimum dan maksimum dari matriks yang akan disiapkan. Berikut adalah isi dari program tersebut.
#include<iostream>
#include<cstdlib>
#include<string>
#include<ctime>
using namespace std;
int main(int argc, char* argv[]){
int rows, columns, min, max;
if (argc==5) {
rows=stoi(argv[1]);
columns=stoi(argv[2]);
min=stoi(argv[3]);
max=stoi(argv[4]);
}
srand(time(0));
int** x=new int* [rows];
for (int i=0; i<rows; i++) {
x[i]=new int[columns];
for (int j=0; j<columns; j++) {
x[i][j]=rand() % (max-min+1) + min;
}
}
for (int i=0; i<rows; i++) {
for (int j=0; j<columns; j++) {
cout << x[i][j] << "\t";
}
cout << endl;
}
free(x);
return 0;
}Kompilasi program di atas dengan perintah g++ -o executablefilename sourcefilename.cpp. Dengan executablefilename adalah berkas yang akan dieksekusi, sedangkan sourcefilename.cpp adalah nama berkas yang berisi program tersebut.
Selanjutnya, jalankan program tersebut dengan perintah ./executablefilename 3 4 1 10, nilai 3 4 1 10 masing-masing adalah:
- jumlah baris matriks
- jumlah kolom matriks
- nilai minimum dari isi matriks
- nilai maksimum dari isi matriks
Gambar 1 berikut mengilustrasikan matriks yang dihasilkan.

Instalasi Geant4 di Ubuntu 22.04
Dari beberapa percobaan, tutorial instalasi yang paling pas ada di sini, yang jadi acuan tulisan ini. Berikut adalah tahapannya.
- Install paket pendukung
- g++
- make
- cmake
- libxerces-c-dev
- libmotif-common
- x11-common
- opengl
- qtcreator
- qtbase5-dev
- qt5-qmake
- build-essential
- checkinstall
- zlib1g-dev
- libssl-dev
- Install CLHEP yang dalam kasus ini adalah rilis 2.4.5.3
- Install Geant4 yang dalam kasus ini adalah rilis 4.11.0.2
Untuk instalasi CLHEP dan Geant4, yang perlu diperhatikan adalah pada setiap directory, perlu dibuat 3 sub directory yang masing-masing adalah build, source dan install. Ekstraksi CLHEP dan Geant4 akan disimpan di sub directory source. Kemudian, instalasi di sub directory install. Sedangkan sub directory build digunakan untuk kompilasi. Sesuaikan struktur directory pada petunjuk sesuai dengan lokasi yang Anda inginkan dan rilis paket yang digunakan.
Kemudian, hal lain yang perlu diperhatikan adalah saat kompilasi Geant4 (khususnya ketika menjalankan perintah make -j 6), ada paket tambahan yang perlu diunduh. Saya baru tahu, perintah make -j 6 adalah proses kompilasi multithread sebanyak 6 thread. Stabilitas koneksi internet menjadi penting karena jumlah dan ukuran paket yang diunduh dan menurun sumber ini, kompilasi secara multithread ini dapat menyebabkan kesalahan muncul ketika proses mengunduh dilakukan. Jika ada kesalahan terkait unduhan tersebut, ulangi saja perintah make -j 6 tersebut. Sayapun perlu setidaknya 5x mengulang. Kira-kira, pesan kesalahan yang muncul akibat gagal unduh tersebut ditunjukkan oleh Gambar 1 berikut. Pesan kesalahan bisa berbeda pada paket yang mengalami gagal unduh. Beruntungnya, pengulangan dilakukan dari posisi terakhir kesalahan terjadi.

Sedangkan Gambar 2 menunjukkan akhir dari proses kompilasi.

Terakhir, Gambar 3 menunjukkan akhir dari tahap instalasi Geant4.

Terakhir, konfigurasi parameter LD_LIBRARY_PATH dengan menjalankan perintah source geant4make.sh. Pada instalasi saya, berkas geant4make.sh berada di /home/arya/Geant4/install/4.11.0.2/share/Geant4-11.0.2/geant4make
Untuk menguji apakah instalasi yang kita lakukan berhasil, kita dapat mencobanya menggunakan contoh kasus yang sudah disediakan. Pada instalasi saya, letaknya ada di /home/arya/Geant4/install/4.11.0.2/share/Geant4-11.0.2/examples. Selanjutnya, mengikuti petunjuk yang disajikan di sini, kita akan masuk ke sub directory basic/B1. Pada sub directory tersebut, jalankan dua perintah berikut, masing-masing cmake dan make. Setelahnya, akan terbentuk berkas executable bernama example1. Ketika dijalankan, muncul jendela seperti Gambar 4 berikut.

Sampai di sini istirahat dulu. Saya benar2 gak tahu apa yang muncul tersebut,he3…
Menjumlahkan matrix dengan CUDA (2)
Pada tulisan sebelumnya, kombinasi jumlah thread pada setiap block tidak mempengaruhi skema indexing. Berbeda halnya ketika ada beberapa thread yang dikelompokkan pada 1 block, sedangkan sejumlah thread lain dikelompokkan pada block yang lain. Padahal, argumen yang diberikan pada fungsi device_add tetap sama yaitu *d_a, *d_b, *d_c. Jika tidak dilakukan pengaturan indeks, hasil penjumlahan hanya berkerja pada sekian elemen pertama dari array. Perhatikan Gambar 1 dan Gambar 2 berikut.
Gambar 1 menunjukkan hasil penjumlahan 12 elemen vektor (baru sadar, ternyata yang dioperasi adalah vektor, matriks 1D) terakhir yang dilakukan oleh CPU, sebaliknya, Gambar 2 menunjukkan hasil penjumlahan 12 elemen vektor terakhir yang dilakukan oleh GPU, lebih tepatnya isi dari variabel d_c. Karena setiap block berisi sejumlah thread, dan tidak ada pengaturan indexing, maka thread pada setiap block melakukan perhitungan pada sejumlah elemen pertama vektor. Sejumlah elemen itulah yang memperbarui isi dari elemen-elemen awal vektor d_c. Akibatnya, ketika d_c ditampilkan, yang nilainya benar hanya elemen awal saja. Untuk memperbaikinya, lakukan beberapa tahapan berikut.
Tambahkan script berikut sebelum memanggil fungsi device_add seperti pada script di bawah ini. Nilai 8 pada numOfThreads adalah asumsi yang bisa disesuaikan dengan kebutuhan. Sedangkan nilai N mengikuti nilai pada script di postingan ini. Usahakan nilai numOfBlocks bulat (habis dibagi).
int numOfThreads=8;
int numOfBlocks=N/numOfThreads;
device_add<<numOfBlocks,numOfThreads>>>(d_a, d_b, d_c);Berikutnya, tambahkan script berikut di dalam fungsi device_add seperti pada script di bawah ini. Perubahan tersebut dilakukan untuk menyesuaikan skema indexing seperti ditunjukkan pada Gambar 3. Dengan begitu, kita dapat melakukan indexing ke elemen mana saja pada vektor sperti diilustrasikan pada Gambar 4.
__global__ void device_add(int *a, int *b, int *c){
int index = threadIdx.x + blockIdx.x * blockDim.x;
c[index] = a[index] + b[index];
}